Les éléments transposables, des parasites génomiques activés par les infections virales
Les éléments transposables sont des séquences d’ADN parasites des génomes, qui ont la propriété de se déplacer et de se multiplier le long des chromosomes. Lorsqu’ils transposent, ils sont à l’origine de mutations qui peuvent être délétères ou source d’innovations génétiques. S’il est connu que certains facteurs comme les stress peuvent déclencher la transposition, la compréhension de la dynamique de ces séquences dans les génomes n’est encore que partielle. Une étude développée par des chercheurs du Laboratoire de Biométrie Biologie Évolutive (LBBE - CNRS/Univ Claude Bernard/Vetagro Sup) et du laboratoire Infections Virales et Pathologie Comparée (IVPC - INRA/Université Lyon 1/EPHE), publiée dans PNAS en juin, montre que les infections virales sont un nouveau facteur à l’origine de la modulation de l’activité des éléments transposables.
Les éléments transposables sont présents dans tous les génomes. Chez les humains, ils en constituent au moins la moitié du génome. Comme toute source de mutation génétique, ils peuvent être à l’origine de maladies, mais aussi à de rares occasions mener à des innovations génétiques, comme la mise en place du placenta. Leur activité pourrait également être impliquée dans les processus de vieillissement ou dans la différenciation neuronale. Différentes voies de contrôle permettent d’inhiber l’activité potentiellement délétère de ces séquences. L’une des plus étudiée à l’heure actuelle repose sur l’interférence ARN : de petites molécules d’ARN, les ARNpi, ciblent les séquences des éléments transposables, ce qui entraîne leur dégradation ou leur inhibition. Cette voie des ARNpi est essentiellement déclenchée dans les gonades, où se trouvent les cellules germinales, ce qui permet de protéger le génome de la génération suivante des mutations dues aux éléments transposables.
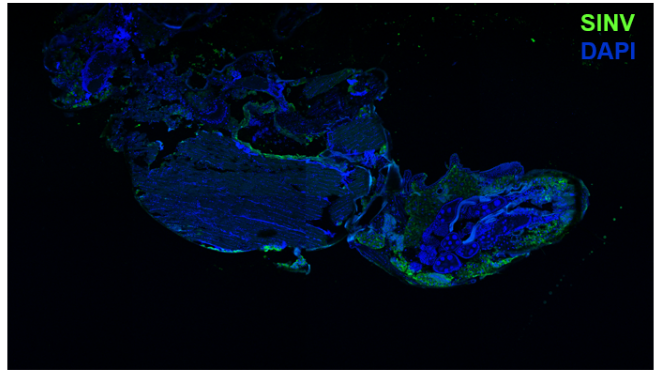
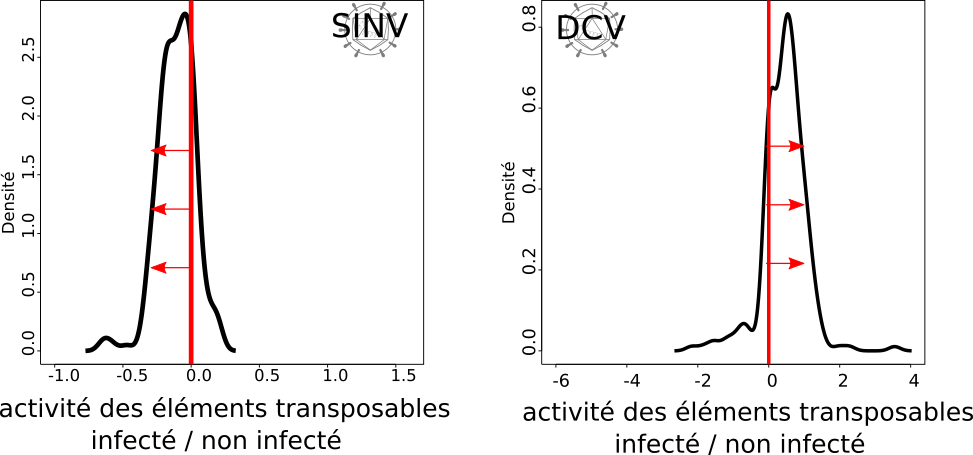
Au niveau somatique, une autre voie d’ARN interférence permet également le contrôle des éléments transposables, par le biais des petits ARNsi. Les voies des ARNpi et ARNsi sont décrites comme indépendantes car elles reposent sur des effecteurs moléculaires distincts. Il se trouve que chez certains organismes, dont les insectes, l’immunité antivirale sollicite également cette voie des ARNsi. En utilisant différentes lignées de drosophile, cette étude montre que l’infection par le virus Sindbis diminue l’activité des éléments transposables dans les tissus somatiques, car la réponse antivirale permet d’augmenter la disponibilité en précurseurs d’ARNpi et ARNsi contre les éléments transposables. Au contraire, certains virus, comme le virus C de la drosophile, codent des suppresseurs de la voie des ARNsi, et ainsi l’infection lève l’inhibition contre les éléments transposables.
Ces résultats suggèrent que les infections virales peuvent moduler l’activité des éléments transposables, ce qui pourrait conduire à des variations de l’intensité de la diversification génétique induite par ces séquences. On peut ainsi proposer que les processus somatiques tels que le vieillissement ou la différenciation neuronale pourraient être affectés. De plus, cette étude montre que si les infections virales n’affectent pas l’activité d’éléments transposables dans les gonades, elles peuvent en revanche moduler les quantités d’ARNpi transmis à la descendance. Les infections virales pourraient ainsi avoir également un impact sur l’évolution des génomes en affectant les taux de mutation.
Référence
Roy, M., Viginier, B., Saint-Michel, E., Arnaud, F., Ratinier, M., Fablet, M. 2020. Viral infection impacts transposable element transcript amounts in Drosophila. PNAS. doi: 10.1073/pnas.2006106117. Epub 2020 May 20.
Equipes de recherche
- Laboratoire de Biométrie Biologie Évolutive (LBBE - CNRS/Univ Claude Bernard/Vetagro Sup)
- Laboratoire Infections Virales et Pathologie Comparée (IVPC - INRA/Université Lyon 1/EPHE)